
 Widok zawartości stron
Widok zawartości stron
Centrum SOLARIS
 Widok zawartości stron
Widok zawartości stron
 Widok zawartości stron
Widok zawartości stron
Strukturalne podstawy rozpoznawania końca transpozonu wyjaśniają centralne cechy systemów transpozycyjnych Tn7
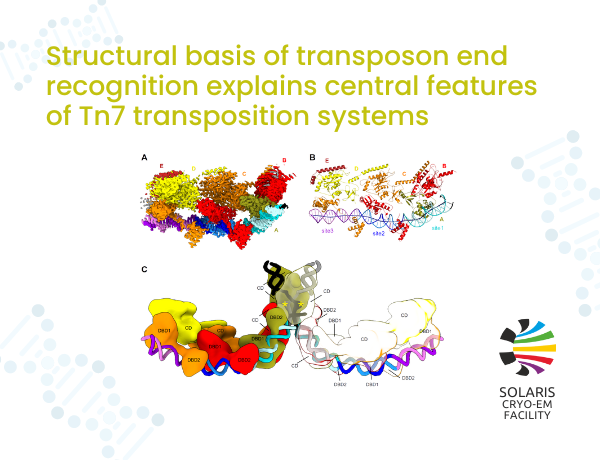
Transpozazy to białka kluczowe dla mobilności transpozonów - elementów genetycznych, które mogą przemieszczać się w obrębie genomów. U prokariotów proces ten może prowadzić do horyzontalnego transferu genów wirulencji, przyczyniając się do powstawania nowych superbakterii. Użytkownicy centrum SOLARIS rozwiązali 2,7 Å strukturę cryo-EM transpozazy TnsB z transpozonu Tn7 oddziałującej z dwuniciowym fragmentem DNA imitującym prawy koniec transpozonu.
Struktura rozwiązana przez zespół naukowców z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie pokazuje, że TnsB wiąże DNA z preferencją sekwencji a nie ze ścisłą specyficznością, oraz że tworzenie macierzy cząsteczek TnsB przekształca tę preferencję w specyficzne rozpoznanie końca transpozonu.
Kriomikroskopia elektronowa w badaniach nad transpozonami
Naukowcy wykorzystali technikę kriomikroskopii elektronowej (cryo-EM) do analizy kompleksów transpozazy TnsB z transpozonu Tn7 z E. coli z dwuniciowymi substratami DNA. W przypadku jednego z substratów, który zawierał trzy miejsca wiążące odpowiadające prawemu końcowi transpozonu, zaobserwowano tworzenie się długich filamentów, które wykorzystano do rozwiązania struktury cryo-EM kompleksu TnsB-DNA (Rysunek 1A, B).
W opublikowanej strukturze domeny cząsteczek TnsB oddziałują z powtarzającymi się miejscami wiązania jak poprzeplatane ze sobą kuleczki na sznurku. Domeny wiążące DNA tworzą tylko kilka specyficznych kontaktów z zasadami azotowymi, co prowadzi do preferencji , a nie ścisłej specyficzności rozpoznawania sekwencji DNA. Tworzenie macierzy cząsteczek TnsB, regulowane przez nakładanie się miejsc wiążących TnsB w prawym końcu elementu Tn7, przekształca tę preferencję w specyficzne rozpoznawanie końca transpozonu. W oparciu o powyższą strukturę naukowcy zaproponowali model przenoszenia nici DNA, w którym końcowa cząsteczka TnsB jest tak przeorganizowana, że jej domena katalityczna znajduje się w pozycji sprzyjającej transpozycji (rysunek 1C).
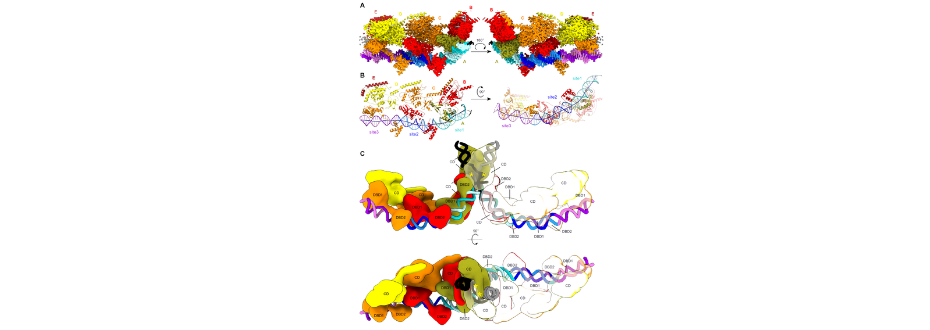
Rys.1. Struktura kompleksu TnsB-DNA.
Nowej generacji narzędzia do edycji
Niektóre niedawno opisane transpozony z szerokiej rodziny Tn7 zawierają zakodowane w elementach systemy CRISPR-Cas, które do wstawienia w docelowe miejsce wykorzystują fragment nici RNA. Wszystkie te systemy kodują transpozazy podobne do TnsB i – ze względu na łatwość programowania miejsca transpozycji - mogą w przyszłości stać się nowej generacji narzędziami do edycji genomów. Nasze analizy strukturalne i biochemiczne transpozazy TnsB oraz zaproponowany model przenoszenia nici DNA zapewniają mechanistyczne zrozumienie, w jaki sposób cząsteczki TnsB mogą interpretować subtelne różnice w sekwencji i położeniu miejsc wiążących i wyjaśniają centralne cechy systemów transpozycji Tn7.
Link do całej publikacji:
Autor: Mariusz Czarnocki-Cieciura
