Widok zawartości stron
Widok zawartości stron
Centrum SOLARIS
 Widok zawartości stron
Widok zawartości stron
 Widok zawartości stron
Widok zawartości stron
Mechanizm działania kluczowego kompleksu białkowego w bakteryjnej rekombinacji homologicznej
Naukowcy z Laboratorium Struktury Białka Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie wyjaśnili mechanizm działania kluczowego kompleksu białkowego w bakteryjnej rekombinacji homologicznej.
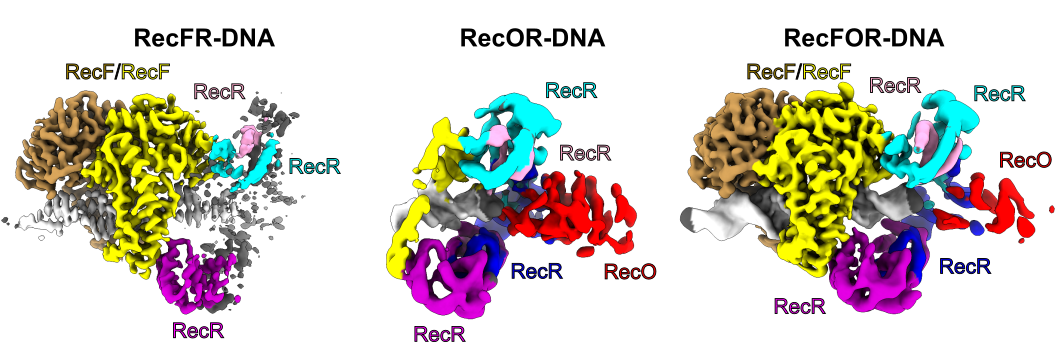
Abstrakt: Rekombinacja homologiczna jest jedną z podstawowych ścieżek naprawy uszkodzonej informacji genetycznej. Polega ona na wytworzeniu jednoniciowej cząsteczki DNA w miejscu uszkodzenia. Następnie, fragment ten jest wykorzystany do poszukiwania matrycy w oparciu o homologię. U bakterii w jedną ze ścieżek naprawy DNA opartych na rekombinacji homologicznej zaangażowane są białka RecF, RecO i RecR. Wiążą się one na styku jednoniciowego (ss) i dwuniciowego (ds) DNA, a następnie ułatwiają wymianę białka SSB, które początkowo pokrywa ssDNA, na białko RecA, które promuje poszukiwanie sekwencji homologicznej. Molekularny mechanizm działania białek RecFOR w tym szlaku był w dużej mierze nieznany. Shivlee Nirwal i współpracownicy z laboratorium prof. Marcina Nowotnego wykorzystali Narodowe Centrum Promieniowania Synchrotronowego SOLARIS do zbadania struktur kompleksów RecF-DNA i RecFOR-DNA oraz ich subkompleksów przy użyciu kriomikroskopii elektronowej, aby wyjaśnić mechanizm współpracy tych białek w rozpoznawaniu połączeń ss-dsDNA.
Rozwinięcie: Struktury subkompleksów RecF-DNA i RecFR-DNA pokazują, jak dimer białka RecF wykorzystuje helikalne wypustki do wiązania dwuniciowej cząsteczki DNA. Struktura subkompleksu RecFR-DNA pokazuje ponadto sposób, w jaki jedna cząsteczka białka RecF oddziałuje z dwoma różnymi regionami pierścienia zbudowanego z czterech kopii białka RecR, co stabilizuje ten pierścień na cząsteczce DNA. Rekonstrukcje o niższej rozdzielczości subkompleksu RecR-RecO oraz całego układu RecFOR-DNA wyjaśniają, w jaki sposób białko RecO jest pozycjonowane, aby oddziaływać z ssDNA i białkiem SSB, co blokuje kompleks na styku ssDNA-dsDNA. Powyższe modele zostały zweryfikowane poprzez szereg eksperymentów biochemicznych i biofizycznych. Podsumowując, uzyskane wyniki integrują i wyjaśniają przekrój danych biochemicznych dostępnych dla systemu RecFOR i zapewniają ramy dla pełnego zrozumienia ścieżki rekombinacji homologicznej RecFOR.
Rysunek 1. Rekonstrukcje kompleksu RecFOR-DNA oraz subkompleksów RecFR-DNA i RecOR-DNA wykonane przy użyciu kriomikroskopii elektronowej.
Napisane przez: Shivlee Nirwal
Link do publikacji: