
 Widok zawartości stron
Widok zawartości stron
Centrum SOLARIS
 Widok zawartości stron
Widok zawartości stron
 Widok zawartości stron
Widok zawartości stron
Struktura białkowego kompleksu Elongator

Karboksymetylacja urydyny w antykodonie tRNA jest niezbędna dla prawidłowej syntezy białek. U archeonów i bakterii wprowadzenie modyfikacji katalizowane jest przez enzym Elp3, natomiast u eukariontów enzym ten wchodzi w skład dużego, wielopodjednostkowego białkowego kompleksu Elongator. W najnowszej pracy naukowej badaczy z Małopolskiego Centrum Biotechnologii (MCB/UJ) opisano pierwszą, wysokorozdzielczą strukturę Elongatora uzyskaną przy pomocy kriomikroskopii elektronowej (cryo-EM), która rzuca światło na rolę podkompleksu Elp456. Wyniki badań zostały opublikowane w tym tygodniu w Nucleic Acids Research jako artykuł przełomowy.
Naukowcy z Grupy Badawczej Maxa Plancka z MCB Uniwersytetu Jagiellońskiego w Krakowie opisali rolę Elp456, mniejszego podkompleksu kompleksu Elongatora, ze szlaku modyfikacji tRNA. Badania grupy dr hab. Sebastiana Glatta zostały przeprowadzone we współpracy z naukowcami z Uniwersytetu w Kassel, Berlińskiego Uniwersytetu Technicznego oraz Europejskiego Laboratorium Biologii Molekularnej w Hamburgu.
Elongator jest dużym i wielopodjednostkowym (Elp1-6) kompleksem białkowym złożonym z dwóch odrębnych podkompleksów: Elp123 i Elp456. Elongator jest odpowiedzialny za katalizę pierwszego etapu modyfikacji 34 urydyny (U34, tzw. wobble uridine), a dokładniej jej węgla w pozycji piątej, poprzez przyłączenie grupy karboksymetylowej (cm5), która to grupa ulega dalszej modyfikacji w wyniku aktywności innych enzymów. W celu zapewnienia efektywnego kotranslacyjnego fałdowania powstającego łańcucha polipeptydowego, tylko w pełni zmodyfikowane tRNA wiąże się w optymalny sposób z rybosomami biorącymi udział w translacji. Zespół dr hab. Glatta przygotował wszystkie próbki w Pracowni Biologii Strukturalnej MCB i zebrał większość danych przy pomocy wysokiej klasy kriomikroskopu elektronowego Titan Krios G3i, który znajduje się w Narodowym Centrum Promieniowania Synchrotronowego SOLARIS. Uzyskane dane posłużyły następnie do określenia struktur przestrzennych kilku stanów pośrednich oraz w pełni uformowanego kompleksu Elongatora z drożdży i myszy.
Uzyskane wyniki wykazały bardzo wysokie zachowanie struktury i funkcji Elongatora wśród eukariontów. Analizy in vitro i in vivo, dopełniły główne ustalenia strukturalne, pozwoliły na szczegółową charakterystykę molekularną dwóch podkompleksów Elongatora i tego, jak oddziałują one ze sobą. „Subtelne różnice strukturalne pomiędzy Elp123 związanym z tRNA a wolnym Elongatorem pozwoliły nam zaproponować funkcję Elp456, która według nas jest związana z uwalnianiem zmodyfikowanych tRNA z podkompleksu katalitycznego.” dodaje dr Marcin Jaciuk, główny autor pracy. O tym, jak ważna jest funkcja Elp456, mówi dr hab. Sebastian Glatt, ostatni i korespondencyjny autor. Stwierdza, że „Porównując centra aktywne drożdżowego i mysiego Elp123 w różnych stanach aktywności, udało nam się zrobić duży, kolejny krok w scharakteryzowaniu molekularnych mechanizmów aktywności modyfikacyjnej Elongatora. Projekt, który rozpocząłem dekadę temu podczas mojego stażu podoktorskiego w EMBL, wreszcie osiągnął swój cel - zrozumienie, jak wygląda i działa ten ważny kompleks białkowy”.
Cała publikacja dostępna pod linkiem:
M. Jaciuk et al., Cryo-EM Structure of the Fully Assembled Elongator Complex, Nucleic Acids Res gkac1232 (2023). doi:10.1093/nar/gkac1232
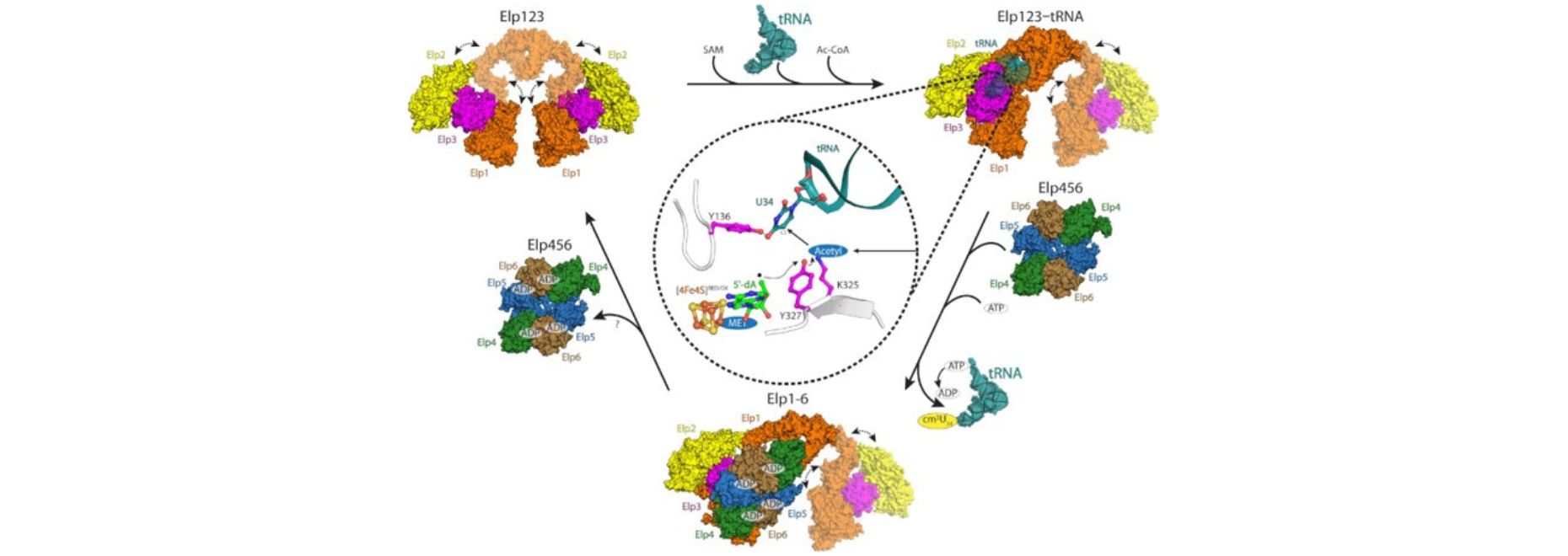
Schemat reakcji Elongatora. Schematyczny przegląd poszczególnych intermediatów reakcji eukariotycznego kompleksu Elongator. U ssaków wyższych wysoce elastyczny Elp123 wiąże substrat tRNA, SAM i Ac-CoA w celu modyfikacji U34. Źródło: Nucleic Acids Res, gkac1232, https://doi.org/10.1093/nar/gkac1232
Praca została wsparta w ramach programu OPUS16 (2018/31/B/NZ1/03559) Narodowego Centrum Nauki, European Research Council w ramach programu Unii Europejskiej Horyzont 2020 oraz programu innowacyjnego (101001394) Fundacji na rzecz Nauki Polskiej (TEAM TECH CORE FACILITY/2017-4/6).
